Ученые смоделировали короткие нуклеотидные
последовательности – аптамеры, – с помощью которых можно почти в десять раз
быстрее, чем методом ПЦР, определять наличие частиц коронавируса в слюне.
Аптамеры специфично связываются с одним из самых редко мутирующих белков
вируса, благодаря чему со 100 % точностью выявляют как его уханьский вариант,
так и штаммы Омикрон и Дельта. Кроме того, авторы описали молекулярные
механизмы взаимодействия вирусных белков с аптамерами. Предложенный подход
поможет в разы ускорить и удешевить тестирование на COVID-19, а также выявлять
заболевание на самых ранних стадиях. Результаты исследования, поддержанного грантом
Президентской программы Российского научного фонда (РНФ), опубликованы в
журнале Molecular Therapy: Nucleic Acid.
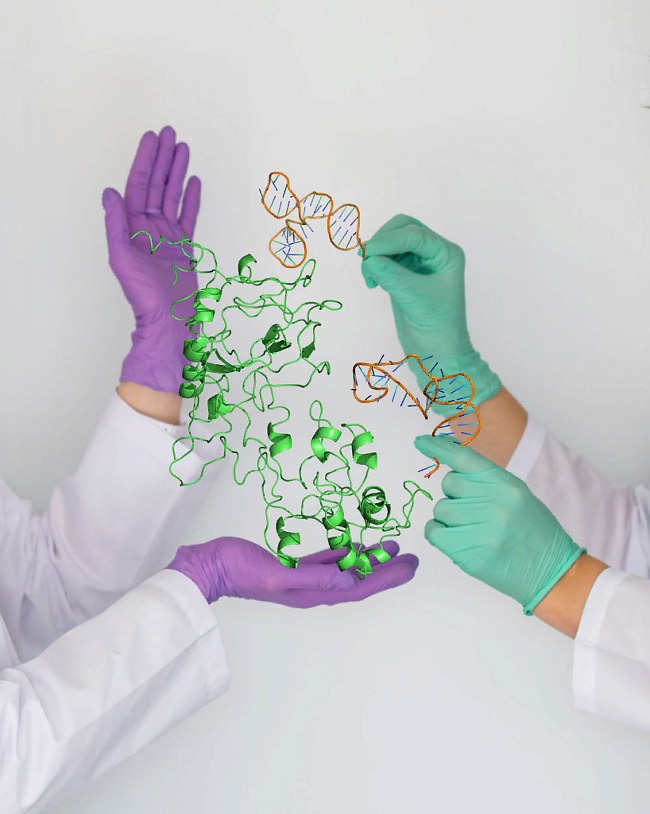
Модель соединения аптамеров с N-белком коронавируса.
Источник: Анна Кичкайло.
Пандемия COVID-19 за три года унесла жизни более 6,8
миллионов человек, при этом вирус был выявлен почти у каждого десятого жителя
планеты. Но далеко не все (особенно в начале пандемии) проходили тестирование
на наличие вируса из-за высокой стоимости и длительного времени анализа. Чтобы
уменьшить риск развития тяжелого течения заболевания, а также замедлить
распространение коронавируса, необходимы быстрые и точные методы диагностики,
которые позволят выявлять инфекцию на самых ранних стадиях. Наиболее часто тест
на COVID-19 проводят с помощью полимеразной цепной реакции (ПЦР), которая
позволяет обнаружить в образцах слизи из носоглотки гены нескольких вирусных
белков. Однако такая процедура довольно дорогая и длительная – анализ занимает
несколько часов.
Уже в первые дни пандемии китайские ученые предложили использовать
для диагностики аптамеры – искусственно синтезированные короткие нуклеотидные
последовательности. Эти молекулы к тому моменту уже использовались при
разработке средств диагностики, лекарств против рака и некоторых наследственных
заболеваний. Аптамеры работают по схожему с антителами принципу: они прочно
связываются с антигеном, в случае COVID-19 – с вирусными белками, поэтому их
можно использовать в качестве «маркеров» для выявления инфекции. Удобной
мишенью для аптамеров служит N-белок в оболочке коронавируса, поскольку в нем
крайне редко происходят мутации, по сравнению, например, с шиповидным S-белком,
который распознается человеческими антителами. Однако до сих пор данный метод не
использовался повсеместно из-за того, что механизмы взаимодействия N-белка и
аптамеров оставались недостаточно понятными, а это не позволяло найти наиболее
эффективные последовательности для связывания с патогеном.

Участники исследовательской группы. Справа налево: Полина
Артюшенко, Ирина Щугорева, Анастасия Рогова. Источник: Анна Кичкайло.
Ученые из Федерального исследовательского центра «Красноярский научный
центр СО РАН» (Красноярск), Красноярского государственного медицинского университета имени
профессора В. Ф. Войно-Ясенецкого (Красноярск) и Сибирского федерального
университета (Красноярск) с коллегами из Канады подобрали такие аптамеры,
которые имеют максимально возможное сродство, то есть способность связываться,
с N-белком коронавируса. Сначала авторы синтезировали множество коротких
цепочек – длиной всего в 60 нуклеотидов – со случайным набором этих «букв».
Затем специальную подложку с закрепленным на ней N-белком на время поместили в
раствор с нуклеотидными последовательностями. Далее подложку промыли, чтобы те
последовательности, которые не прикрепились к N-белку, смылись, а связавшиеся с
ним – остались. Процедуру повторили несколько раз, чтобы удалить те молекулы,
которые слабо взаимодействовали с белком. В результате исследователи получили
16 аптамеров с наибольшим сродством к N-белку коронавируса.
Затем авторы проверили чувствительность и силу связывания аптамеров с целевым белком
с помощью специального датчика, отслеживающего процесс их взаимодействия по
электрическим характеристикам. Наилучшие результаты показали три молекулы,
обозначенные как tNSP1, tNSP2 и tNSP3. Чтобы доказать, что аптамеры
избирательно распознают только SARS-CoV-2, ученые провели аналогичные опыты с
N-белком другого коронавируса – MERS. Все варианты связывали его примерно на
50% хуже, что, с одной стороны, говорит об их высокой избирательности
(поскольку связалась только половина молекул), а с другой – указывает на
родство SARS-CoV-2 и MERS, а также на схожесть в структуре их белков. Однако
этого оказалось достаточно для того, чтобы однозначно различить эти вирусы.
По сочетанию двух признаков – силы связывания и избирательности – авторы
выбрали лучший аптамер, которым оказался tNSP3. Далее его использовали для
обнаружения коронавируса в образцах человеческой слюны, содержащей уханьский
вариант коронавируса, а также варианты Дельта и Омикрон. Во всех случаях tNSP3
позволил определить N-белок в количествах, сопоставимых с чувствительностью
ПЦР. При этом предложенный подход оказался почти в десять раз быстрее и
дешевле.
«Вклад российской стороны в эту разработку очень важен,
мы проанализировали механизмы связывания и рассчитали энергии взаимодействия
аптамеров с белками коронавируса. Благодаря этому нам удалось усовершенствовать
методику тестирования COVID-19 с помощью аптамеров. При использовании в
клинической практике этот метод поможет ускорить проведение анализа. Однако в
ближайшее время нам предстоит еще сравнить точность данного подхода с ПЦР, для
чего потребуется масштабное исследование на большем количестве пациентов. Это
уже наша вторая работа по созданию аптамеров к коронавирусу. В прошлой
публикации, поддержанной этим же грантом РНФ, мы смоделировали с
помощью суперкомпьютеров Межведомственного суперкомпьютерного центра аптамеры к
S-белку, которые сейчас исследуются на наличие противовирусного эффекта», –
рассказывает руководитель проекта, поддержанного грантом РНФ, Анна
Кичкайло, доктор биологических наук, ведущий научный сотрудник и заведующая
лабораторией цифровых управляемых лекарств и тераностики Федерального
исследовательского центра «Красноярский научный центр СО РАН».
Источник: пресс-служба РНФ.